Metodi di
analisi genetica
Nelle analisi genetiche possono essere utilizzate le seguenti metodiche:
- Esame cromosomico standard: è una vera e propria fotografia del DNA per verificare che il numero delle strutture che lo contengono (cromosomi) sia corretto. Questo numero è caratteristico di ogni specie: quella umana possiede 46 cromosomi organizzati in 23 coppie. I primi 44 cromosomi formano 22 coppie dette “omologhe” perché ogni cromosoma è ricevuto da ciascun genitore. La 23esima coppia è formata dai cromosomi sessuali, che sono ereditati in modo diverso dagli omologhi. Un individuo femmina eredita 2 cromosomi X, ognuno proveniente da un genitore. Un individuo maschio, invece, eredita il cromosoma Y paterno ed il cromosoma X dalla madre. Esistono alcune malattie genetiche causate da anomalie nel numero e/o nella struttura dei cromosomi. Quando il medico sospetta queste condizioni, è possibile eseguire l’esame cromosomico standard per confermarle o per escluderle. E’ importante ricordare che questa tecnica può identificare soltanto anomalie di numero piuttosto grandi, come la mancanza o l’eccesso di interi cromosomi o di loro porzioni rilevanti. (Figura 2)
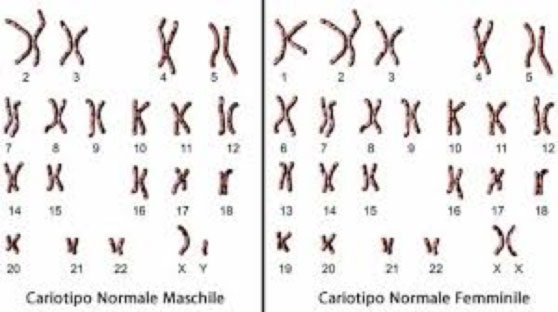
Figura 2: Cariotipo umano normale maschile e femminile (23 geni)
- Sanger: questa tecnica prende il nome dal genetista che la scoprì, nel 1977. La tecnica si basa sulle proprietà strutturali del DNA: esso è una lunga catena formata da milioni di anelli: ogni anello si chiama “nucleotide”. Il sequenziamento di Sanger è la lettura (manuale o automatica) del DNA ovvero l’insieme di nucleotidi letti nel giusto ordine. Nel DNA sono presenti 4 tipi di nucleotidi, contenenti una delle basi: A, G, C, T. Come è approfondito nel sito https://www.emanuelemicaglio.it/category/articolo/ A e G sono purine, C e T sono pirimidine. Se vogliamo determinare la sequenza di un frammento di DNA da 100 nucleotidi, significa che vogliamo comprendere quali sono i 100 nucleotidi nell’ordine esatto. Alla fine del sequenziamento si ottiene un insieme di lettere, es. AAGTGTGTCTGTCAGTTCTAGAACC…, apparentemente prive di significato, in realtà a seconda del test che si sta eseguendo avranno un proprio significato. In altre parole, il risultato di un sequenziamento di Sanger consente di stabilire se e di quale mutazione un paziente sia portatore. Maggiori informazioni possono essere ricavate dal link https://www.emanuelemicaglio.it/2019/02/18/la-genetica-medica-storia-di-un-futuro-remoto/.
- Array-CGH: è un miglioramento tecnologico dell’esame cromosomico standard che consente di rilevare anomalie cromosomiche di numero con dimensioni molto ridotte. Queste possono provocare, come quelle più grandi, una mancanza o un eccesso di DNA che pertanto non avrà più una concentrazione normale nelle cellule del paziente. La tecnica del CGH si basa sulla frammentazione del DNA del paziente che viene confrontato con quello di un soggetto sicuramente sano di uguale sesso. Alcune cardiopatie possono essere causate dalla mancanza di specifici frammenti nei cromosomi 21, 22 o sul cromosoma X. Riconoscere precocemente queste anomalie consente da un lato di trattare la cardiopatia e dall’altro di prevenire le possibili complicanze.
- Next Generation Sequencing (NGS): è una recente semplificazione della tecnologia Sanger in uso dal 2012 e basato sulla lettura multipla e computerizzata del DNA. Consente di studiare non più un gene per volta ma un “pannello” di geni, comprendente decine di frammenti di DNA, ognuno dei quali può venire letto centinaia e centinaia di volte. In Cardiogenetica è comune che la stessa malattia possa essere causata da molte differenti mutazioni, in geni diversi. Pertanto la tecnologia NGS ha rivoluzionato la nostra branca, consentendo in molti casi di ottenere la conferma o l’esclusione di un sospetto formulato su base clinica.
- MLPA: è un esame di approfondimento che si utilizza in genere quando il sequenziamento (Sanger o NGS) risulta negativo ma clinicamente esiste il sospetto fondato di una condizione genetica. La tecnica è simile a quella del CGH e consente di verificare se il gene (= frammento di DNA) oggetto del test sia presente in due copie. In caso contrario possono verificarsi due eventualità: nessuna copia (= delezione genica) o tre copie (= duplicazione genica). In entrambe questi casi, la conseguenza per il paziente può essere una cardiopatia. E’ da segnalare che un meccanismo come la perdita o l’acquisto di un gene può provocare cardiopatie complesse ed in genere sindromiche (ossia con coinvolgimento di altri organi oltre il cuore).
- Whole Genoma Sequencing (WGS): permette di esaminare l’intero patrimonio genetico di un paziente ed al momento (settembre 2019) ha un impiego limitato alla ricerca. L’esame si basa sulla stessa tecnica dell’NGS soltanto che non limita il campo d’indagine, comprendendo anche regioni di DNA non ancora studiate (Figura 3). Il nostro gruppo analizza i campioni con metodica WGS soltanto nei pazienti affetti dalla sindrome di Brugada. Pertanto il test studia sia i geni oggi associati alla sindrome di Brugada che tutti gli altri. Pertanto i pazienti che aderiscono al protocollo di ricerca vengono accuratamente informati sulle possibili implicazioni dei risultati per la loro salute. Ogni paziente può decidere di sapere soltanto la causa della propria condizione oppure di conoscere anche eventuali risultati inattesi, che potrebbero avere un’enorme rilevanza per la loro salute.

Figura 3: schema semplificato dei legami chimici tra i due filamenti del DNA genomico
